【導入事例 Vol.26】
理化学研究所 理学博士 岩根 敦子 様

■経歴
昭和34年1⽉29⽇⽣
○昭和59年3⽉ 東京薬科⼤学薬学研究科修了
○平成19年11⽉〜現在 ⼤阪⼤学⼤学院⽣命機能研究科、医学部(兼任)准教授
○平成25年4⽉〜平成30年3⽉ 理化学研究所 ⽣命システム研究センター ユニットリーダー
○平成30年4⽉〜令和2年3⽉ 理化学研究所 ⽣命機能科学研究センター ユニットリーダー
○令和2年4⽉〜 同 チームリーダー(予定)
○令和元年4月〜現在 広島大学大学院統合生命科学研究科、特任教授
■先生の研究内容について教えてください。

細胞内超微細構造を電⼦顕微鏡や光学顕微鏡など特殊な顕微鏡を使って明らかにする研究しています。⼈間の⽬はミリメートル単位のものしか観察できませんよね。⾍眼鏡だとプランクトン、光学顕微鏡は細胞レベル、電⼦顕微鏡はナノレベルと、⾁眼では⾒ることができない物が拡⼤され、物⾃体の構造が明らかになります。サイズ感でいうと、地球がもし1メートルだと仮定すると、髪の⽑は⼤⼭の⾼さ、細胞は戦艦⼤和、DNAは500円⽟ぐらいでしょうか。光学顕微鏡は照明に光を⽤いますが、電⼦顕微鏡は電⼦線を⽤います。電⼦線を使⽤することで散乱する電⼦を映像化し、細胞⼩器官と呼ばれる超微細な構造の観察を可能にしました。また、タンパク質分⼦をオングストロームの分解能で⾒ることもでき、細胞内超微細構造を観察することで、薬の開発に役⽴ったり、病気の原因を調べたり、⽣き物の命の仕組みを分⼦レベルで探索することにもつながっています。ここのラボには、ミリメートルからナノメートルレベルまで⾒ることができる電⼦顕微鏡や光学顕微鏡がすべて揃っています。⼀つのラボにこれだけの機器が揃っているのは、中国・四国地域でもここだけだと思います。
■FIB-SEM-Tomographyという顕微鏡もあるのですよね。

電⼦顕微鏡のデメリットの⼀つとして、動態が追えないというのがあります。必ず化学固定や物理固定をして、構造をしっかり固めなくては⾒ることができません。通常、電⼦顕微鏡の研究は⽣き物の中から細胞を抽出したり、タンパク質を精製した後、固定をしてキレイになったものの構造を⾼分解能観察するのが⼀般的です。ノーベル賞を取ったクライオ電⼦顕微鏡はタンパク質をコーティングなしで直接観察することができます。私たちが使っているFIB-SEM-Tomographyは光学顕微鏡だけでは観察できない細胞内の標的超分⼦や⼩器官とそれを取り巻く環境を適切な固定や染⾊をして、⽣きた状態に限りなく近い状態を保持しながら⾼分解能で三次元観察をしています。
■電⼦顕微鏡で⾒るための環境作りが⼤切になるのですね。

そうです。電⼦顕微鏡で対象物を⾼真空環境下で観察するには、まず⽔分の取り除かれた試料が必要です。⾃分が⾒たいものを⾒えるようにする染⾊技術や加⼯技術がとても⼤切なのです。FIB(集束イオンビーム)とSEMが同⼀チャンバーに取り付けられたDualBeam装置を使⽤して、イオンでナノメーターレベルに削っています。1000枚、2000枚と連続的かつ、安定的に画像を取得。1枚毎の絵をつなぎ合わせて3次元に再構築して、⽴体にしていくのです。⽴体にするために、観察したあとAmiraという専⽤ソフトを使って解析しています。このソフトは3D画像処理するために⼤容量のメモリを使⽤するのですが、それにあうようなレベルのパソコンということで、アプライドさんのパソコンを導⼊しました。細胞内の標的物とそれを取り巻く環境を丸ごと観察することで、例えば疾患の治療戦略のために有⽤な情報を得ることができます。
■具体的にはどのような研究になるのですか。
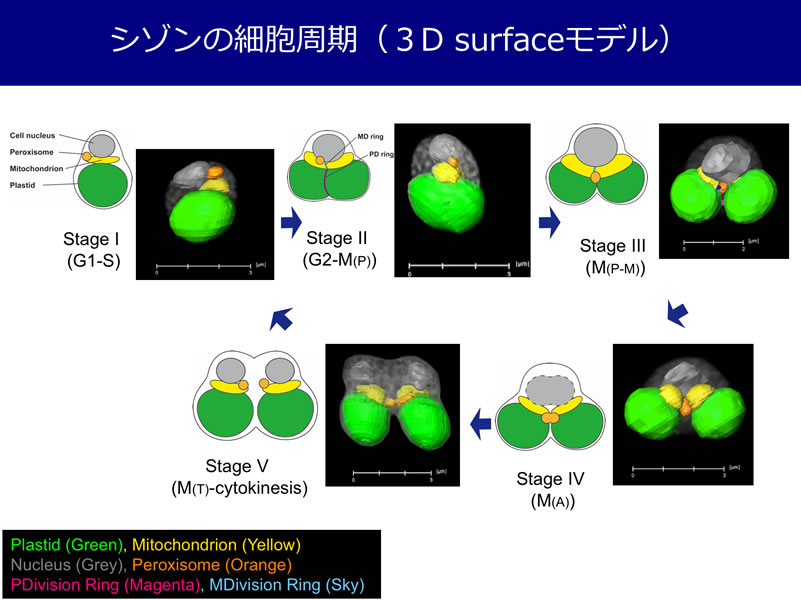
シアニディオシゾンという単細胞の紅藻がイタリアナポリの温泉地で生息しています。日本の草津温泉にもその近緑種が生息しており、28憶年前から⽣き続けている最も原始的な真核⽣物です。オルガネラ(細胞⼩器官)の構成もシンプルで、細胞核、葉緑体、ミトコンドリア、ゴルジ体、ペルオキシソームなどがすべて⼀つずつ含まれています。私たちはこの紅藻の一つの親細胞が確実に二つの娘細胞に細胞分裂する過程に注目。FIB-SEM-Tomographyによる電⼦顕微鏡解析技術の開発や、未だ謎の多い、異なるオルガネラの相互作⽤について研究しています。
現在は地域を活性化するという総務省のプログラムもあり、広島ならではのテーマも多く、広島大学病院の腎臓内科の先⽣とコラボレーションして患者さんの⽣検を3D解析したり、酒類総合研究所さんとアルコール発酵時の⽇本酒の酵⺟のオルガネラの変化過程を電⼦顕微鏡で解析して、構造的な変化を調べるなど、分野も多岐にわたっています。その中でも特に力を入れているのがCTスキャン検査のダメージ評価の判定。CTスキャンは私たちの病気の原因や進⾏度に有⽤な情報を提供してくれますが、放射線をあびることで、体は相当なダメージを受けています。そのダメージも⼈によって差(家族性の差)があるのではないかと⾔われています。動物細胞に放射線照射すると、10分後にはミトコンドリアが癒着したり、内膜がボロボロになったり、核膜にダメージを受けたりするのですが、1時間後にリカバリーしていることも分かってきました。オルガネラがどのように形を変えて⽣きているのかを構造から解明するのに3Dで解析しています。
■3D解析と1枚絵の差はどこにあるのでしょうか。
通常電子顕微鏡は1枚の絵しか見ることができません。例えば、二本指を立てているような画面があったとします。実際はウサギが手足を上げているのですが、投影しているので人間の手にしか見えない。まるごと生き物の構造を見ることで、生命現象を理解することができます。一つの構造モデルができると、様々な方向から観察することができるのも3Dのメリットです。
遺伝子バンクがあるように、タンパク質の世界にもPDB(プロテインデータバンク)というタンパク質の構造を登録しているバンクがあります。世界にはまだまだ確認や認識されていないタンパク質がたくさんあります。タンパク質同士がどのように相互作用して、ATPエネルギーを使ってどうやって動いているのかという研究も行っています。
■ラボでは試料から解析まで一連のことができるのですね。

ゲノム編集や遺伝⼦組換えなどから試料を作って、観察、解析までできるようなラボは世界中探してもほとんどないと思います。そのぐらいすごい機材がここには集まっています。
細胞⽣物の分野の研究で他にはC2C12細胞を分化させて、ある条件にすると筋⾁細胞になるか、脂肪細胞になるかが分かれるのが分かってきていますが、力学的な刺激を与えると、筋肉細胞への分化が促進されるんです。私たちは細胞のどのあたりから筋節(サルコメア)構造ができてきて、広がっているのかを3Dで解析する研究を⾏っています。サルコメアは細いアクチンフィラメントと太いミオシンフィラメントが重なって配列しています。例えば腕を曲げるとき、筋組織を作っているサルコメアが⼀気にATPを加⽔分解して縮みます。ATPは⽣物の⽣命維持活動や運動に必須のエネルギー源で、紅藻の場合は光合成をしてATPを創り出しています。動物細胞は光合成をしているわけではないので、ミトコンドリアがATPを産出します。⼀⼈の成⼈男性が⼀⽇どのぐらいのATPを創り出しているかというと、約60キログラムなんですよ。動いたり、考え事をして消費しているので体重が増えないんです。⼈間が死ぬとATPは新たに作られないので、徐々に死後硬直していきます。
■新たに取り組まれていることはあるのですか?

解析ソフトのAmiraですが、現在は⼈の⼿を使ってミトコンドリアは⻩⾊、核はグレー、⾊素体は緑など区分けをしています。オルガネラを一つずつしか持たない紅藻のようなシンプルな細胞の画像解析に、ベテランの方でも約4時間はかかります。現在広島⼤学⼯学部情報⼯学専攻の栗⽥多喜夫先⽣にAIの技術で膨⼤な画像からオルガネラ抽出ができるようお願いしています。AIだと1分で何⼗、何百という画像が抽出できる。ただし、最後の5%は結局⼈間の⼿で修正しないとダメなのですけどね。





























